Jeder Liter Meerwasser enthält Erbmaterial verschiedenster Lebewesen. Die Biologin Silke Laakmann schafft mit ihrem Team die Grundlagen dafür, aus den DNA-Spuren auf die biologische Vielfalt in einem Meeresgebiet schließen zu können. Dafür kombinieren die Forschenden traditionelle Methoden der Artenbestimmung mit neuen genetischen Verfahren.
Das Naturschutzgebiet Sylter Außenriff ist für Nordsee-Verhältnisse ungewöhnlich vielfältig: Auf dem Meeresboden wechseln sich größere Steinfelder mit Flächen aus Kies und Grobsand ab, dazwischen liegen flache Sandbänke. Die Steine bieten einen festen Untergrund für bunte Riff-Lebensgemeinschaften aus Tieren wie Seenelken, Muscheln, Moostierchen und Schwämmen, während die Sandbänke verschiedene Arten von Schlangensternen und Würmern beherbergen. Im Wasser fühlen sich zahlreiche Fischarten wohl, etwa Dorsche, Sprotten oder Heringe, aber auch Plattfische und die seltenen Flussneunaugen. Das gute Nahrungsangebot zieht wiederum Meeressäuger an – Schweinswale und Seehunde.
Um einen Überblick über die Biodiversität in Meeresschutzgebieten wie dem Sylter Außenriff zu bekommen, müssen Umweltforschende bislang einen hohen Aufwand betreiben: Sie werfen Netze mit verschiedenen Maschenweiten aus, um Wasserbewohner unterschiedlicher Größen zu fangen, nehmen Proben vom Meeresboden oder setzen Unterwasserkameras aus, die Fotos und Filmaufnahmen vom Untergrund machen.
Doch es geht auch anders: „Wir brauchen eigentlich nur eine Wasserprobe“, sagt die Biologin Dr. Silke Laakmann. Die Forscherin und ihr Team machen es sich zunutze, dass im Meerwasser ein stark verdünntes Sammelsurium biologischer Hinterlassenschaften herumschwimmt: Zellreste, Schleim, Schuppen, Haare oder auch Fäkalien sowie Überreste verwesender Tiere. Auch wenn diese Mischung wenig appetitlich klingt, kann das genetische Material darin – richtig analysiert – erstaunliche Einblicke in die biologische Vielfalt in einem Meeresgebiet liefern. Fachleute sprechen von Umwelt-DNA oder kurz eDNA (Englisch: environmental DNA).

An dem großen Ziel, die Biodiversität im Meer allein anhand von Wasserproben zuverlässig erfassen zu können, arbeitet Laakmann am Helmholtz-Institut für Funktionelle Marine Biodiversität an der Universität Oldenburg (HIFMB). Das Institut wurde 2017 als institutionelle Kooperation zwischen der Universität und dem Alfred-Wegener-Institut, Helmholtz-Zentrum für Polar- und Meeresforschung (AWI) gegründet. „Wir untersuchen, ob die Ergebnisse, die uns die eDNA liefert, auch der Wirklichkeit entsprechen – ob wir also wirklich alle vorkommenden Tiere einer Region erfassen“, sagt die Forscherin, die am HIFMB seit 2018 die Fokusgruppe Marine Molekularökologie leitet. Das Institut beherbergt derzeit zwei dieser Fokusgruppen. Sie sind ein Instrument, um vielversprechende junge Wissenschaftlerinnen und Wissenschaftler gezielt zu fördern und gleichzeitig besonders innovative Forschungsrichtungen am HIFMB zu verankern.
Im Trend ist auch das Verfahren, das im Zentrum der Arbeit von Laakmann und ihrem fünfköpfigen Team steht. Die Analyse der eDNA hat sich in den vergangenen Jahren zu einem mächtigen neuen Werkzeug der Umweltwissenschaften entwickelt. Das Potenzial der Methode ist groß – weil sie es ermöglicht, komplette Artengemeinschaften egal welcher Größe auf einmal zu analysieren, und viele neue Einblicke in die Artenvielfalt liefert. Zudem sind weniger massive Eingriffe in die Umwelt nötig als bei traditionellen Probenahmemethoden, was insbesondere für Meeresschutzgebiete wichtig ist.
Im Verbundprojekt CREATE, geleitet vom Oldenburger Biodiversitätsexperten und HIFMB-Direktor Prof. Dr. Helmut Hillebrand, spielt Umwelt-DNA eine zentrale Rolle, um die Biodiversität in einzelnen Meeresschutzgebieten sowie deren Verbindung miteinander zu analysieren. Die Europäische Auster wird dabei als Beispielart verwendet. „Eine wichtige Frage ist, ob sich die Austernlarven vom Schutzgebiet Borkum Riffgrund aus mit den Strömungen in andere Meeresgebiete ausbreiten, in denen sie bisher nicht angesiedelt wurden“, erläutert Laakmann. Innerhalb des Projekts, das durch die Deutsche Allianz Meeresforschung vom Bundesforschungsministerium gefördert wird, bauen die Biologin und ihr Team derzeit ein eDNA-Archiv für die Nordsee auf, anhand dessen sich der aktuelle Stand der Biodiversität und die Auswirkungen zukünftiger Umweltveränderungen dokumentieren lassen sollen. Der Umweltwissenschaftler Dr. Kingsly Chuo Beng, Postdoktorand in der Fokusgruppe und Experte für eDNA-Untersuchungen, leitet das entsprechende Arbeitspaket.
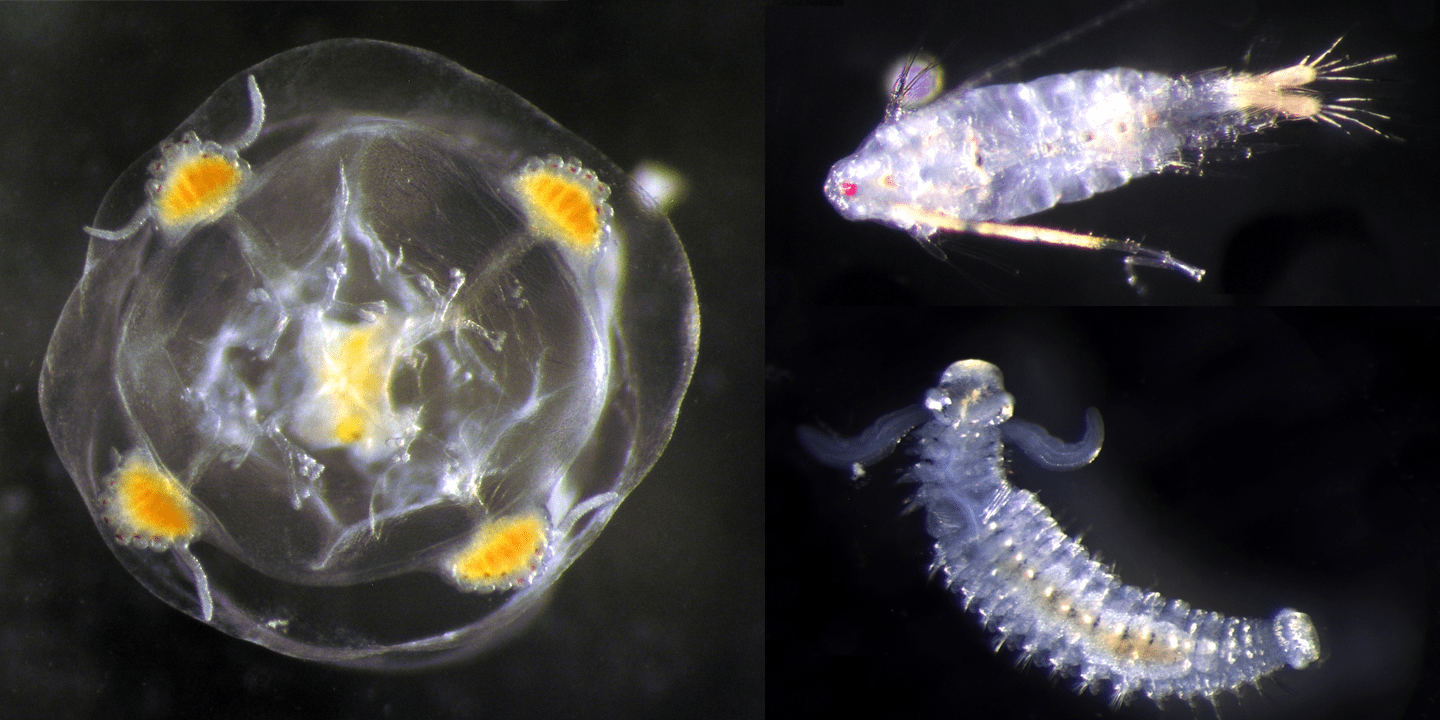
Die Grundlagen dafür haben die Forschenden in Laakmanns Gruppe in den vergangenen vier Jahren gelegt. „Seit einigen Jahren findet ein Umbruch in der Biodiversitätsforschung statt, sozusagen von den klassischen morphologischen zu molekularen Methoden der Artenbestimmung“, berichtet die Forscherin. Man könnte auch sagen: vom Mikroskop zum Gen. Sie selbst ist noch in den traditionellen Verfahren geschult. „Während meiner Doktorarbeit an der Universität Bremen und meiner Zeit als Postdoktorandin am Forschungszentrum Senckenberg am Meer in Wilhelmshaven habe ich sehr viel Zeit im Sortierlabor verbracht und die Tiere unter dem Binokularmikroskop anhand ihrer äußeren Form bestimmt“, erinnert sie sich. Ihr Spezialgebiet ist tierisches Plankton, in der Fachsprache Zooplankton genannt, insbesondere die vielfältige Gruppe der winzigen Ruderfußkrebse.

Foto: Uni Oldenburg
Um die neuen genetischen Methoden in Zukunft routinemäßig zum Umweltmonitoring einsetzen zu können, findet derzeit ein Abgleich mit den traditionellen Verfahren statt. Laakmann und ihr Team widmen sich vor allem dem Zooplankton in der Nord- und Ostsee, untersuchen aber auch Material aus Patagonien, der Arktis und Südafrika. Mehr als 3.000 Proben haben sie bereits gesammelt, die künftig im neuen HIFMB-Gebäude archiviert werden sollen.
Um einen Überblick über die Biodiversität zu erhalten, setzen sie meist drei verschiedene Verfahren ein: Sie sammeln nach wie vor Plankton auf traditionelle Weise mit langen, dünnen Netzen. An Bord kommt dabei meist eine bräunliche, flockige Masse aus stecknadelkopf- bis wenige Millimeter großen Tierchen. „Diese Proben werden geteilt“, berichtet Laakmann. Die Arten in der einen Hälfte bestimmen entweder sie und ihre Teammitglieder weiterhin klassisch unter dem Binokularmikroskop – oder Dr. Astrid Cornils, eine Kollegin vom AWI, mittels halbautomatischer Bildanalyse. Die andere Hälfte des Planktons wird zu Brei verarbeitet und anschließend genetisch analysiert. Das dritte Verfahren ist die Analyse der Umwelt-DNA. Dafür wird das Erbmaterial aus Wasserproben extrahiert, die aus verschiedenen Wassertiefen stammen.
Die molekularbiologischen Methoden zur Artenbestimmung gleichen ansatzweise denen, die derzeit in Corona-Testlabors bei PCR-Tests zum Einsatz kommen: Es werden charakteristische Bereiche der Gene vervielfältigt und anschließend sequenziert, also ausgelesen.
„Mir ist es wichtig, eine Verbindung zu den Organismen zu behalten.“
Silke Laakmann
Die Umweltwissenschaften konzentrieren sich dabei zumeist auf einen Genabschnitt mit dem Namen COI (Cytochrom-c-Oxidase Untereinheit I). „Die Sequenz dieses speziellen Genfragments ist für jede Art einzigartig“, erläutert Laakmann. In riesigen Datenbanken inventarisieren Forschende unterschiedlicher Initiativen weltweit Lebewesen anhand des genetischen Codes ihres COI-Gens. Ähnlich wie Lebensmittelverpackungen im Supermarkt durch einen Strichcode (englisch: barcode) gekennzeichnet sind, lässt sich jede Tierart, die einmal in einer Datenbank registriert ist, anhand ihres genetischen Codes identifizieren. Laakmann selbst hat in ihrer Zeit als Doktorandin und Postdoktorandin dazu beigetragen, diese Bibliothek des Lebens um zahlreiche Arten zu erweitern, darunter frei im Wasser und am Boden lebenden Tiere der Nordsee sowie Ruderfußkrebse aus der Arktis, der Antarktis und der Tiefsee.
Wird das in einer Umweltprobe enthaltene riesige Sammelsurium an Erbgut parallel analysiert – ein Vorgang, bei dem Tausende unterschiedlicher DNA-Sequenzen entstehen – sprechen Fachleute von „Metabarcoding“.
„In unseren Proben haben wir Millionen dieser Gen-Schnipsel von allen möglichen Tieren. Nach der Sequenzierung vergleichen wir diese Gensequenzen mit den Einträgen in Datenbanken, sodass wir am Ende eine Liste von Arten oder Tiergruppen erhalten“, erläutert Laakmann das Verfahren. In den vergangenen vier Jahren haben die Forschenden eine Art Werkzeugkasten für den Umgang mit der eDNA entwickelt. „Es ging um Fragen wie: Wie viel Wasser brauchen wir, um möglichst viele Arten in einem Gebiet zu erfassen? Wie häufig müssen wir Proben nehmen? Wie soll der Filter aussehen? Welche Datenbanken verwenden wir?“, berichtet die Biologin. Auch mit Schwellenwerten und Rechenalgorithmen befasste sich das Team. Mit den Resultaten ist sie zufrieden: „Wir wissen jetzt, dass wir die verschiedenen Gruppen von Wirbellosen und Fischen, die in der Nordsee vorkommen, mit dem eDNA-Metabarcoding tatsächlich finden. Und wir verstehen ganz gut, wie wir die Methode bei unterschiedlichen Fragestellungen bestmöglich einsetzen können.“
Um das Vorkommen bestimmter Arten in großen Naturschutzgebieten wie Borkum Riffgrund oder Sylter Außenriff zu erforschen, sei es beispielsweise am besten, an vielen verschiedenen Orten Wasserproben zu nehmen. In einer Studie zu Bodenlebewesen, bei der die Forschenden mit einem Team des AWI zusammenarbeiteten, das Langzeitbeobachtungen in Meeresschutzgebieten durchführt, kombinierten sie dagegen eDNA-Analysen mit Sedimentproben und Unterwasserfotos. In besonders dynamischen Meeresgebieten könne es wiederum nötig sein, tagelang immer wieder an derselben Stelle Wasserproben zu nehmen, um alle vorkommenden Arten zu erfassen.

Foto: HIFMB | Kingsly Chuo Beng
Für Laakmann ist es wichtig, sich auch in Zukunft nicht nur auf die genetischen Verfahren zu verlassen. „Ich möchte sogenannte integrative Methoden nutzen, also klassische und neue Methoden kombinieren. Denn dann bekommen wir das Beste aus beiden Welten.“ Als Meeresbiologin möchte sie es nicht missen, winzige Flohkrebse, Hydromedusen oder Schwebgarnelen unter dem Binokular zu bestimmen. „Ich finde das wichtig, um eine Verbindung zu den Organismen zu bekommen, die ich untersuche“, betont sie – ein Faktor, der fehle, wenn man nur abstrakt mit Gensequenzen arbeitet.

Foto: HIFMB | Nora Bendig
Darüber hinaus ist die Interpretation der Ergebnisse des eDNA-Metabarcodings oft nicht ganz einfach. Laakmann kontrolliert daher alle Artenlisten daraufhin, ob die Resultate Sinn ergeben. Wenn etwa in einer Liste eine unerwartete Zooplankton-Art auftaucht, prüft die Forscherin gegebenenfalls sogar, wie vertrauenswürdig die Einträge in den Datenbanken sind. „In einem Fall haben wir festgestellt, dass ein seltsames Muster in den Daten auf einer falschen Identifizierung nach traditioneller Methode beruhte“, erzählt sie.
Dass in den Artenlisten immer wieder Landtiere wie Huhn, Rind, Wildschwein oder die Waldmaus auftauchen, ist eher ein Kuriosum. „Teilweise können wir das sogar erklären“, sagt Laakmann: Genetisches Material von landwirtschaftlich genutzten Tieren wie Hühnern und Rindern werde womöglich durch Flüsse eingetragen, ebenso wie Hinterlassenschaften von Wildschweinen, die an der Küste leben. Ballastwasser sei eine weitere mögliche Quelle exotischen Erbguts. „Die Waldmaus ist uns bislang allerdings ein Rätsel“, schmunzelt die Biologin, die diesem Problem zusammen mit Kingsly Chuo Beng nun auf den Grund gehen will.
„Wir sehen erstmals, welche Larven wann im Wasser treiben.“
Silke Laakmann
Hin und wieder entdecken die Forschenden auch sogenannte kryptische Arten – Spezies, die genetisch neu sind, aber äußerlich einer anderen, bereits bekannten Art gleichen. Zudem steigen die Chancen, seltene oder bedrohte Tiere aufzuspüren, darunter auch die verschiedenen Meeressäuger. „Wir finden mit den neuen genetischen Methoden mehr Arten als vorher“, betont Laakmann. All das belege das große Potenzial der Umwelt-DNA: Sie macht bislang verborgene Bereiche der Biodiversität zugänglich.
Bei den Larven im Zooplankton, die von Bodenlebewesen und Fischen stammen, eröffnet sich sogar eine völlig neue Welt: Oft lassen sich diese winzigen Wesen mit traditionellen Bestimmungsmethoden nur grob verschiedenen Tiergruppen zuordnen – etwa den Seeigeln oder den Muscheln. Per Metabarcoding können Forschende jedoch problemlos viele dieser Larven bis zur Art bestimmen – vorausgesetzt, das entsprechende Tier ist bereits in einer Datenbank inventarisiert. „Jetzt sehen wir erstmals, welche Larven zu welcher Jahreszeit im Wasser treiben, und können daraus Rückschlüsse über Vorkommen und Fortpflanzungszyklen ziehen“, berichtet Laakmann. Besonders im späten Frühjahr und im Sommer, wenn sich viele Tiere fortpflanzen, ergibt die eDNA-Analyse teils viermal so viele Arten in Zooplanktonproben wie die traditionelle Bestimmung. Auch die Larven der Europäischen Auster lassen sich mit dem Binokular kaum identifizieren: Die weniger als einen Millimeter großen freischwimmenden Stadien – rundliche Klumpen, die sich mit lappenartigen Fortsätzen fortbewegen – sehen genauso aus wie die aller anderen Muscheln. Laakmann und ihr Team sind dennoch zuversichtlich, die Reisewege des Weichtier-Nachwuchses in den Weiten der Nordsee aufdecken zu können: In Voruntersuchungen für das Projekt CREATE waren sie bereits erfolgreich dabei, das Erbmaterial der Larven in Wasserproben nachzuweisen.
Ute Kehse (Uni Oldenburg)
Dieser Artikel ist ursprünglich erschienen im Einblicke-Magazin der Universität Oldenburg. Hier die ganze Ausgabe lesen.